ワサビの染色体レベルでのゲノム解読に成功
本学の応用生物科学部山根京子准教授および学部四年生山本祥平氏(研究当時)、東京工業大学生命理工学院伊藤武彦教授および田中裕之研究員、学部四年生堀立樹氏(研究当時)、情報・システム研究機構国立遺伝学研究所豊田敦特任教授、東京都立大学矢野健太郎客員教授の研究グループは、世界に先駆けてワサビのハプロタイプ )1 レベルでの高精度な全染色体参照ゲノム )2 解読に成功しました。
ワサビは日本原産の香辛野菜であり、日本食文化に欠かせない重要な食材です。近年の和食ブームの影響もあり世界的な需要も増加するなかで、抗酸化作用や癌抑制作用など、機能性食品としても注目されています。今回私たちはワサビの全ゲノムの高精度な解読に成功しました。用いた植物材料は、全国わさび品評会において常に上位入賞し、現在最も市場価値が高い品種'真妻(まづま)'です(図1)。本研究では、次世代シーケンサー(PacBioおよびIllumina,)とHi-Cとよばれる染色体立体配座捕捉法を用いて染色体スケールのアセンブリを行いました。その結果、ワサビは7本の染色体が4組からなる異質四倍体生物であることを明らかにするとともに、7本x4組=合計28本の染色体から構成される合計1,512.1 Mbの配列データを明らかにしました。どのくらい長い配列をつなげられたかを示す指標であるN50の長さは55.67 Mbでした。さらに、リードマッピングと系統解析により、28本の染色体を二組のサブゲノム、更にはそれぞれを二組のハプロタイプの割り当てに成功しました。三種類の方法(Benchmarking Universal Single-Copy Orthologs、Merqury、Inspector)で評価した結果、得られたゲノム配列は高品質で完全性の高いものであることが示されました。今回明らかとなったゲノム配列は、遺伝や進化などの基礎研究、品種改良など農業分野、さらには在来や野生ワサビの保全のための情報整備など、多くの分野での活用が期待されます。
本研究成果は、日本時間2023年7月11日にNature姉妹誌Scientific Dataのオンライン版で発表されました。
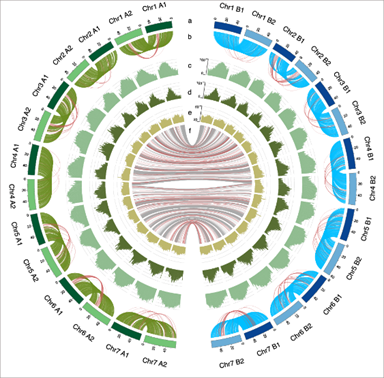
ワサビゲノムの特徴
発表のポイント
- 日本を代表する香辛料であるワサビのゲノム配列を染色体レベルにまで繋ぐことに成功し、ハプロタイプレベルで高精度に解読しました
- 本ゲノム情報はワサビの辛味成分の進化機構の解明、栽培化の起源、品種改良など、遺伝資源としての基盤情報の構築に役立つことが期待されます
詳しい研究内容について
論文情報
- 雑誌名:Scientific Data
- 論文名:Haplotype-resolved, chromosomal-level assembly of wasabi (Eutrema japonicum) genome
- 著 者:Hiroyuki Tanakaa, Tatsuki Horia, Shohei Yamamotob, Atsushi Toyodac, Kentaro Yanod, Kyoko Yamaneb*, Takehiko Itoha* (*責任著者)
- 所 属:
[a] 東京工業大学生命理工学院、[b] 岐阜大学応用生物科学部、[c] 情報システム研究機構国立遺伝学研究所、[d] 東京都立大学 - DOI番号:10.1038/s41597-023-02356-z
- 論文公開URL:
 https://www.nature.com/articles/s41597-023-02356-z
https://www.nature.com/articles/s41597-023-02356-z
用語解説
- 1) ハプロタイプレベル: (今回用いている意味としては)相同染色体を片親由来ずつに区別して
- 2) ゲノム: 生物種がもつ遺伝情報の総体











