工学部化学・生命工学科 喜多村徳昭助教,マクロライド系抗生物質の簡単な全合成経路を報告 (2016.5.19付Natureへ掲載)

このたび,本学工学部化学・生命工学科 喜多村徳昭助教が米・ハーバード大学留学中に関わった研究の成果が,2016年5月19日付のNatureに掲載されましたのでお知らせします。喜多村助教は岐阜大学工業倶楽部(工学部同窓会)による工学振興基金を受けて渡航され,留学期間は2013年4月~2014年3月です。
本研究成果は,米・ハーバード大学Andrew G. Myers教授らが開発したマクロライド系抗生物質の実用的な全合成経路を報告したものであります。従来の方法よりも非常に容易に合成ができるだけでなく,その収量も格段に増やせることから,彼らの手法は今後の新しい抗生物質の開発に大いに役立つものであります。
抗生物質は,細菌等の増殖や機能を阻害する化合物として私たちの生活に欠かせない存在であります。細菌の抗生物質への耐性(薬剤耐性)の拡大は深刻な問題であり,これまで以上に効果的な治療薬の開発が急務となっています。現在使用されているマクロライド系抗生物質(抗菌薬)の多くは,天然由来物質の部分修飾(半合成:構造の一部分について少しずつ合成を行う)によって作られており,その過程は非常に複雑であり,合成できる抗生物質はごく少量であります。単純な構造の低分子化合物から一つひとつ作り上げる方法(全合成)も提唱されていますが,リニア合成法(直線型合成法)ではステップ数の多さ,得られる量ともに半合成と同様の課題を持ち,目的とする抗生物質以外の合成は容易ではありませんでした。

Myers教授らの研究グループでは,従来の方法より単純かつ柔軟な過程を経たマクロライド系抗生物質のコンバージェント合成法(収束的合成法:モジュラーアプローチ)を開発しました。この方法は,合成できる目的物の量を飛躍的に増加させるのみならず,多様な構造をもつ抗生物質の合成への応用が可能で,非常に優れた合成方法ということができます。実際に,Myers教授らは,300種類以上のマクロライド系抗生物質を合成し,それらの抗菌活性を検証しました。
収束的合成の鍵となるのは「ビルディングブロック」です。目的とする抗生物質を得るための効率的な合成経路を決定(逆合成解析)し,合成可能な単位となる化合物(モジュール)を選定します。これをビルディングブロックと呼びます。ビルディングブロックを合成する過程と,それらを組み合わせる過程とに分け,目的である抗生物質を合成します。
本学の喜多村助教は,主にこのビルディングブロック(全8種類のうち3種類)の作成に貢献しました。
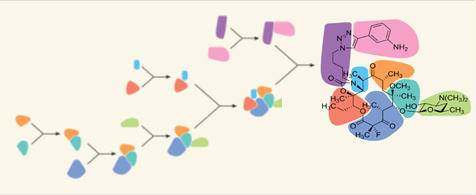
小さいブロックを組み合わせて自動車を作成する場合を考えてみます。従来のリニア合成法では,例えば自動車の底の部分から作る場合は,まず底を作成し,その底に少しずつブロックを足して最終的に車を形作るイメージです。何度もブロックを足して作成するため,どうしても煩雑な過程となります。また,目的とする車の種類が変わった場合,スタート地点である「底の作成」からやり直す必要があります。
これに対して今回の方法では,自動車の底,エンジン,天井,ドアといったパーツをあらかじめ用意し,それらを組み合わせることで自動車を作成するイメージです。従来の方法に対して,シンプルな過程となり,また,違う形状の車を作りたい場合でも,エンジンはそのままで,天井やドアといったボディについて違うパーツを準備すれば,作成が可能です。この例の中での"パーツ"1つ1つが,本学喜多村助教が作成に貢献した「ビルディングブロック」に相当します。
本手法により合成したマクロライド系抗生物質を様々な病原性細菌に対して評価したところ,大半の化合物が抗菌活性を持っており,既存のマクロライド系抗生物質に耐性を持つ菌株に対しても強い効果を示すものもありました。
これにより,今までマクロライド系抗生物質の使用が難しかったケースや,細菌耐性が生まれてしまったケースにおいても,使用が可能になるなどの効果が見込めます。また,今回開発された抗生物質の合成方法は,他の抗生物質の合成にも応用できるものであり,効率的な抗生物質の開発に繋がることが期待されます。
発表論文
論文タイトル
A platform for the discovery of new macrolide antibiotics
著者
Ian B. Seiple1*†, Ziyang Zhang1*, Pavol Jakubec1, Audrey Langlois-Mercier1†, Peter M. Wright1†, Daniel T. Hog1†, Kazuo Yabu1†,Senkara Rao Allu1, Takehiro Fukuzaki1, Peter N. Carlsen1, Yoshiaki Kitamura1†, Xiang Zhou1, Matthew L. Condakes1†,Filip T. Szczypiński1†, William D. Green1† & Andrew G. Myers1 1Department of Chemistry and Chemical Biology, Harvard University, Cambridge, Massachusetts 02138, USA. †Present addresses: Department of Pharmaceutical Chemistry, University of California, San Francisco, California 94158, USA (I.B.S.); Novartis Pharma AG, Chemical and Analytical Development, CH-4002 Basel, Switzerland (A.L.-M.); McKinsey and Company, 55 East 52nd Street, 21st Floor, New York, New York 10022, USA (P.M.W.); Bayer Pharma AG, Medicinal Chemistry, Müllerstrasse 178, 13353 Berlin, Germany (D.T.H.); Medicinal Chemistry Research Laboratories, Daiichi Sankyo Co., Ltd, Shinagawa R&D Center, 1-2-58 Hiromachi, Shinagawa, Tokyo 140-8710, Japan (K.Y.); Department of Chemistry and Biomolecular Science, Gifu University,1-1 Yanagido, Gifu 501-1193, Japan (Y.K.); Department of Chemistry, University of California, Berkeley, California 94720, USA (M.L.C.); Department of Chemistry, University of Cambridge, Lensfield Road, Cambridge CB2 1EW, UK (F.T.S.); Trinity College, University of Cambridge, Cambridge CB2 1TQ, UK (W.D.G.). *These authors contributed equally to this work.
掲載誌:
Nature (Published : 19 May 2016| doi:10.1038/narute 17967)











